Un equipo de la Universidad Goethe de Frankfurt clasifica todas las E3 ligasas humanas, abriendo nuevas posibilidades para terapias dirigidas a través de la degradación de proteínas.
Las células constantemente crean nuevas proteínas y descomponen las que ya no necesitan. Para este proceso de descomposición, utilizan principalmente el sistema ubiquitina-proteasoma (UPS), donde las proteínas se marcan con ubiquitina y se dirigen a un “desguace”, el proteasoma. En este sistema, la enzima E3 ligasa juega un papel central, actuando como un “intermediario” que facilita el marcado de las proteínas destinadas a la degradación. Un equipo de investigación de la Universidad Goethe de Frankfurt ha creado ahora, por primera vez, un catálogo sistemático de todas las E3 ligasas humanas y sus interrelaciones, identificando 40 de ellas como posibles actores en nuevas clases de fármacos, como los PROTAC, que podrían utilizarse en terapias contra el cáncer.
Mantener el orden es un gran desafío logístico para la célula: una sola célula de mamífero contiene miles de millones de moléculas de proteína que deben formarse, utilizarse y descomponerse con precisión. Para la degradación a través del sistema ubiquitina-proteasoma (UPS), ciertas proteínas se adornan con cadenas de múltiples moléculas de ubiquitina y luego son trituradas por un proteasoma. El paso crucial en este proceso es la selección de la proteína objetivo específica: las E3 ligasas son enzimas que actúan como “intermediarios” moleculares, uniéndose a la proteína objetivo y luego mediando la transferencia de ubiquitina de una enzima E2 a la proteína objetivo.
Dado que cada E3 ligasa solo reconoce un número limitado de proteínas objetivo, las células poseen un gran y diverso repertorio de E3 ligasas. Un equipo de investigación de la Universidad Goethe de Frankfurt, liderado por el Dr. Ramachandra M. Bhaskara del Instituto de Bioquímica II, ha compilado ahora a todos los miembros de esta “familia de intermediarios” en un catálogo y ha demostrado, por primera vez, cómo las E3 ligasas humanas se relacionan entre sí y qué significa esto para su función, el reconocimiento de las proteínas objetivo y el desarrollo de fármacos.
Un mapa basado en datos del “E3-Ligoma”
Para describir esta parentela de intermediarios –el llamado “E3-Ligoma”–, los investigadores compararon las características de las E3 ligasas entre sí utilizando ordenadores y con la ayuda de la inteligencia artificial. Posteriormente, confirmaron los resultados en experimentos de cultivo celular y pudieron extraer conclusiones sobre la función de las E3 ligasas. A través de estas funciones, definieron 13 familias principales, así como varias subfamilias que reflejan mejor las similitudes entre los miembros de las E3 ligasas que las simples similitudes de secuencia de aminoácidos o estructura. Bhaskara explica: “Nuestro enfoque basado en datos y aprendizaje automático revela funciones específicas de las familias de E3 ligasas. Por ejemplo, los miembros de una familia son importantes para los programas de reparación del ADN y la prevención de la muerte celular no deseada, mientras que otros participan en la defensa antiviral”.
Además de su papel en la degradación de proteínas, las E3 ligasas también participan en procesos de transducción de señales. Para ello, se utiliza ubiquitina que no se utiliza para la degradación de proteínas. Esto demuestra que las E3 ligasas son importantes para numerosas vías de señalización celular y mecanismos de enfermedad.
Importancia para los terapéuticos de nueva generación
El nuevo mapa de E3 ligasas es particularmente relevante para los nuevos fármacos como los PROTAC, que permiten la degradación selectiva de proteínas. Los PROTAC (Quimeras de Dirigimiento de Proteólisis) son moléculas bifuncionales que acercan una E3 ligasa a una proteína relevante para la enfermedad, asegurando que la proteína se marque con ubiquitina y se destruya mediante un proteasoma. Aunque el campo de investigación de los PROTAC se está desarrollando rápidamente, la mayoría de los PROTAC existentes se basan en un pequeño número de E3 ligasas bien caracterizadas.
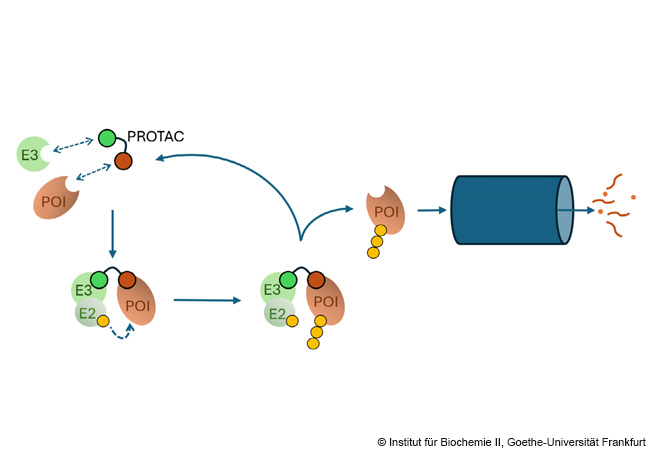
A través del análisis sistemático de todo el E3-Ligoma, el equipo de investigación de Frankfurt identificó 40 E3 ligasas adicionales que podrían ser potencialmente adecuadas para el desarrollo de nuevos PROTAC. Especialmente importante: el mapa de E3 ligasas permite utilizar o adaptar socios de unión ya establecidos de E3 ligasas para E3 ligasas relacionadas y transferir principios de diseño. De esta manera, el desarrollo de nuevos PROTAC para más tejidos corporales, constelaciones celulares y enfermedades podría acelerarse.
Recurso abierto para la comunidad investigadora
Dado que numerosos grupos de investigación en todo el mundo están trabajando en enfoques para la degradación selectiva de proteínas, el equipo de la Universidad Goethe ha puesto a disposición pública el E3-Ligoma completo a través de una base de datos especializada, para que otros investigadores puedan basarse en la clasificación y los conocimientos funcionales.
Publicación: Arghya Dutta, Alberto Cristiani, Siddhanta V. Nikte, Jonathan Eisert, Yves Matthess, Borna Markusic, Cosmin Tudose, Chiara Becht, Varun Jayeshkumar Shah, Thorsten Mosler, Koraljka Husnjak, Ivan Dikic, Manuel Kaulich, Ramachandra M. Bhaskara: Multi-scale classification decodes the complexity of the human E3 ligome. Nature Communications (2025)

